Comparación de protocolos moleculares para la detección del Virus del fruto rugoso marrón del jitomate en hospedantes solanáceos
por
Erika J. Zamora Macorra,
Katia Aviña Padilla*,
Rosemarie W Hammond,
Daniel L. Ochoa Martínez
*Autor de Correspondencia.
Recibido: 23/8/2023 – Publicado: 23/12/2023 – DOI: https://doi.org/10.18781/R.MEX.FIT.2023-5
Resumen Antecedentes/Objetivo. El virus del fruto rugoso marrón del jitomate (ToBRFV) ha surgido como una amenaza significativa para los cultivos de la familia Solanaceae, incluidos el tomate y el pimiento. Su presencia en México desde 2018 ha generado preocupación sobre su impacto en la producción agrícola. La detección temprana y precisa de este patógeno es crucial para prevenir su propagación y mitigar sus efectos. En México, se emplean varias técnicas moleculares para su diagnóstico, incluyendo RT-PCR convencional, RT-qPCR y RT-qPCR multiplex.
Materiales y Métodos: El objetivo de esta investigación fue evaluar la eficiencia de diferentes métodos de extracción de ARN en combinación con oligonucléotidos PCR específicos para la detección de ToBRFV.
Resultados. Entre los métodos probados, el protocolo de extracción de ARN CTAB-Trizol combinado con PCR anidada utilizando oligonucleótidos reportados por Dovas et al. (2004) se identificó como el método molecular más sensible para detectar el virus.
Conclusión. Este hallazgo destaca la importancia de seleccionar la combinación adecuada de protocolos de extracción y amplificación para lograr la sensibilidad y precisión óptimas en la detección de ToBRFV. Palabras clave: Virus del fruto rugoso marrón del jitomate, ToBRFV, cultivos de Solanaceae, extracción de ARN, RT-PCR, detección molecular, producción agrícola.
Palabras clave:
Virus del fruto rugoso marrón del jitomate, ToBRFV, cultivos de Solanaceae, extracción de ARN, RT-PCR, detección molecular, producción agrícola

Figura 1. A-C) En plantas de jitomate saladette recolectadas en invernaderos, se observaron síntomas evidentes de mosaico, deformación foliar y estrechamiento de las hojas. Estas plantas dieron positivo para el virus del fruto rugoso marrón del jitomate (ToBRFV). D-E) Las observaciones visuales en hojas inoculadas de Nicotiana revelaron la presencia de lesiones cloróticas y necróticas localizadas causadas por la infección con ToBRFV. Las plantas utilizadas para el estudio y que resultaron positivas al virus provinieron de Tecomán, Colima, México.
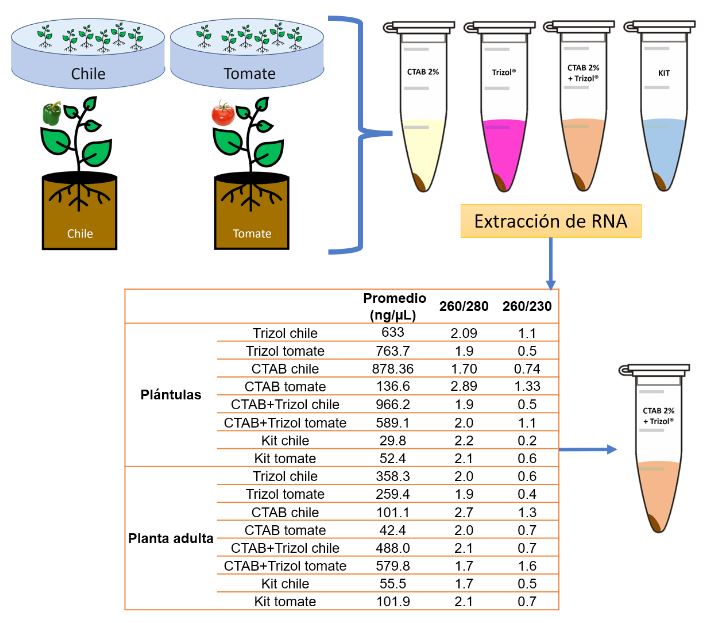
Figura 2. La evaluación de los métodos de extracción de ARN total implicó la comparación de su eficacia y rendimiento, medidos a través de lecturas de absorbancia en un espectrofotómetro Nanodrop 2000®. De los métodos probados, que incluyeron Trizol®, CTAB 2 % y CTAB 2 %-Trizol®, el protocolo CTAB 2 %-Trizol® demostró producir la concentración más elevada de ARN total, mientras que el kit de aislamiento de ARN resultó en la concentración más baja. Estos datos fueron registrados mediante mediciones de absorbancia a longitudes de onda de 260/280 y 260/230. El Nanodrop 2000® se utilizó para cuantificar la concentración de ARN extraído y evaluar su calidad a través de estas relaciones de absorbancia. Los resultados señalan que el protocolo CTAB 2 %-Trizol® fue especialmente efectivo en la extracción de ARN de alta calidad de las muestras de plantas, lo que lo convierte en una opción adecuada para análisis moleculares posteriores
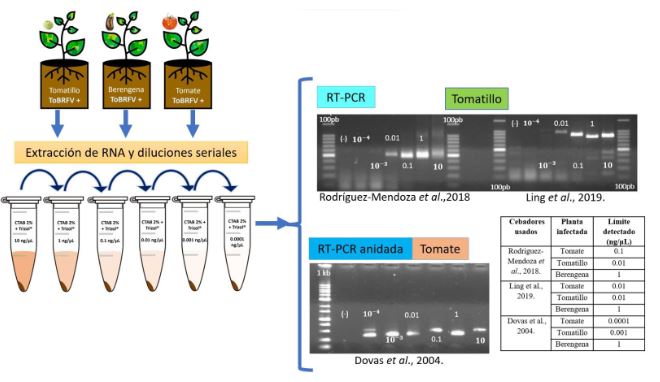
Figura 3. Evaluación y sensibilidad de los oligonucleótidos PCR. Análisis electroforético en geles de agarosa al 1.5 % de los productos de RT-PCR y RT-PCR anidada (tamaño esperado de los oligonucleótidos de Ling 842 pb; tamaño esperado de los oligonucleótidos de Rodríguez-Mendoza 475 pb; tamaño esperado de los oligonucleótidos de Dovas 400 pb). (-): agua esterilizada en lugar de ARN. 100pb= 100pb DNA Ladder (Invitrogen®). 1Kb= 1000 pb DNA ladder (Promega®). 10-3 y 10-4 = 0.001 y 0.0001 ng μL-1
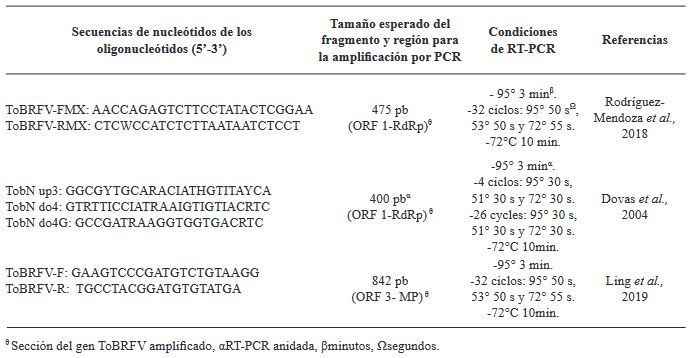
Cuadro 1. Oligonucleótidos probados en este estudio para la detección del ToBRFV en jitomate, tomatillo y berenjena
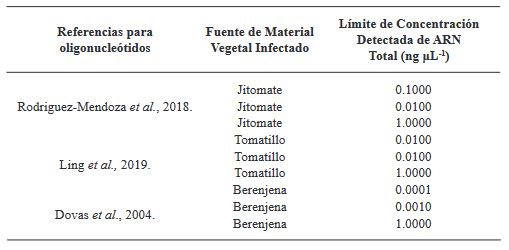
Cuadro 2. Comparación de referencias de oligonucleótidos, fuentes vegetales y límites de detección de ARN en la fuente de material vegetal infectado.

